Rのメタアナリシス用のパッケージmetaについて、以前の投稿で紹介しました「Copilotに聞きながらメタアナリシスを実行:metaパッケージを使って」
今回は、Excelで各研究から研究ID、メタアナリシスに必要なデータを抽出した表を作成し、そのデータからRでmetaを動かして、メタアナリシスを実行するR用のスクリプトを作成してみました。Inverse variance methodによるランダム効果モデルで、研究間の分散はREML(Restricted Maximum Likelihood)法を用いています。対応する効果指標のタイプはRR, RD, OR, HR, MD, SMDです。
Excelで作成する表は、研究ID、人数、平均値、ハザード比の自然対数などのデータと、labelのカラムに、研究IDのカラムのラベル、対照、介入の名称、アウトカム、効果指標のタイプと省略形、フォレストプロットのレイアウト(meta, RevMan5, JAMAのいずれか)、Prediction Intervalを含めるかどうか(No Prediction IntervalまたはPrediction Interval)を入力します。フォレストプロットはこれらのラベルを表示します。サンプルのExcelファイルはこちらからダウンロードできますので、右クリックしてダウンロードし、データを書き換えて使用してください。
Rを起動しておいて、新規エディタのフィールドに以下のスクリプトを貼り付け、カーソルをその行に置きます。
exdat = read.table("clipboard", sep = "\t", header = TRUE);source("https://stat.zanet.biz/useRs/scripts/with-meta-all.R")Excelのシートを開き、データを含む範囲を選択して、コピー(Ctr+C)操作を実行し、Rにもどり、上記の一行を実行します。必要なスクリプトをウエブサーバーから読み込み、実行して、メタアナリシスの結果がコンソールに出力され、Forest plotとFunnel plotが作成されます。
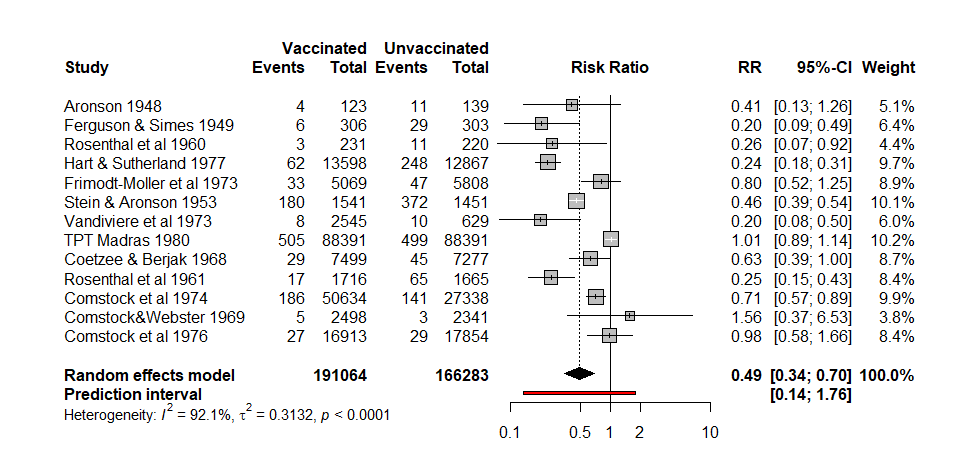
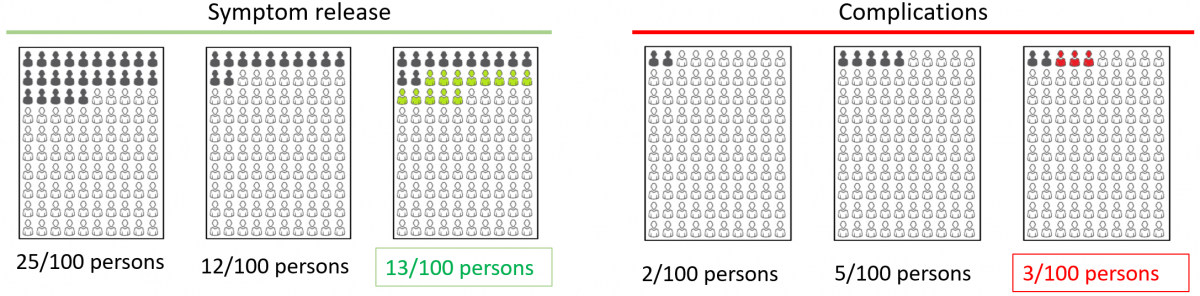
さて、Mindsの評価シートでメタアナリシスに必要なデータを入力してある場合、左上セルA3(診療ガイドライン)から、右下の方の効果指標(種類)のカラムで、データを含む行までの範囲を選択して、コピー(Ctr+C)操作を実行し、Rにもどり、以下のコードのexdato=read.delim(“clipboard”,sep=”\t”,header=TRUE);を実行します。これにより、クリップボード経由で、データが変数exdatoに格納されます。そして、###Startの行から、#####Endの行までを選択し(反転した状態で)、実行します(Windowsの場合は実行ボタンをクリックするか、Ctr+R、Macはcommand+enterを押します)。メタアナリシスが実行され結果がコンソールに出力され、Forest plotとFunnel plotが作成されます。なお、クリップボードからデータを読み込む操作と、#####Start以下のスクリプトは分けて実行してください。
#Meta-analysis with meta from Minds sheet.
#Select the range A3 ~ down the effect measure column and run these scripts in R.
#Read data via clipboard from Minds sheet.
exdato=read.delim("clipboard",sep="\t",header=TRUE);
#####Start
#Reformat the data frame.
source("https://stat.zanet.biz/useRs/scripts/to_forest_metafor.R");
#Add cells to the label clumn for layout and Prediction Interval.
if(nrow(exdat)==6){
new_row <- exdat[1, ]
new_row[,] <- NA
exdat <- rbind(exdat, new_row)
}
if(nrow(exdat)==7){
new_row <- exdat[1, ]
new_row[,] <- NA
exdat <- rbind(exdat, new_row)
}
#Choose a forest plot layout: remove # of the format you want to use.
exdat$label[7] = "meta"
#exdat$label[7] = "RevMan5"
#exdat$label[7] = "JAMA"
#Choose "No Prediction Interval" or "Prediction Interval": remove # of the what you want.
exdat$label[8] = "No Prediction Interval"
#exdat$label[8] = "Prediction Interval"
#Do meta-analysis with meta to create forest plot and funnel plot.
source("https://stat.zanet.biz/useRs/scripts/with-meta-all.R")
#####EndこのスクリプトではForest plotの形式は3種類から選択する必要があり、#のついていない行の形式になります。デフォルトではmetaの形式になっていますので、例えばもしRevMan5の形式にしたければ、#exdat$label[7] = “meta”と#を行頭に付け、exdat$label[7] = “RevMan5″とこちらの#は外してください。
Prediction Intervalを含めるかどうかも同じように、行頭に#が付いていない方が実行されます。デフォルトではNo Prediction Intervalになっています。もしPrediction Intervalを含めたいのであれば、#exdat$label[8] = “No Prediction Interval”と行頭に#を付け、exdat$label[8] = “Prediction Interval”とこちらの#は外してください。
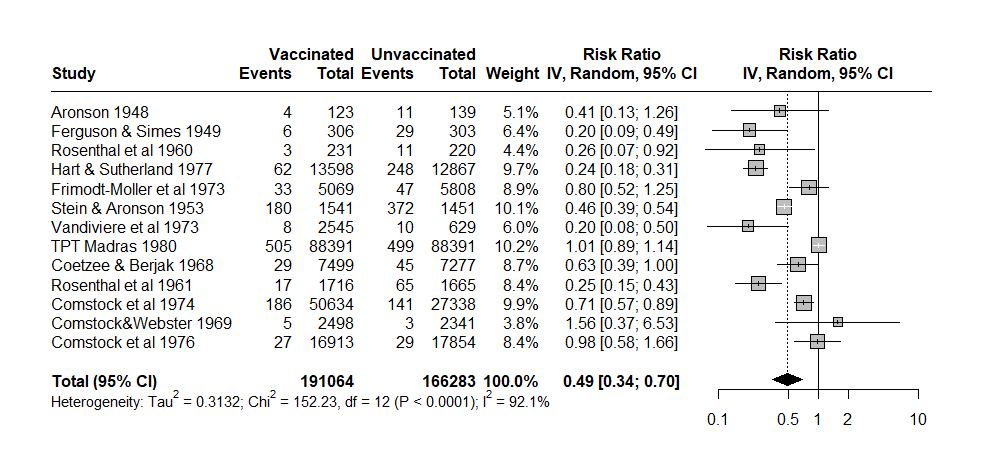
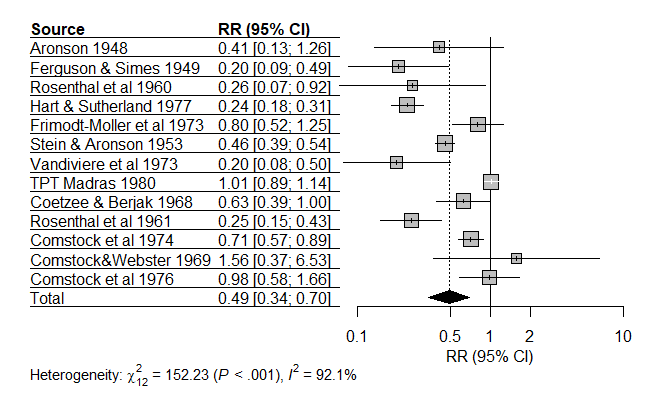
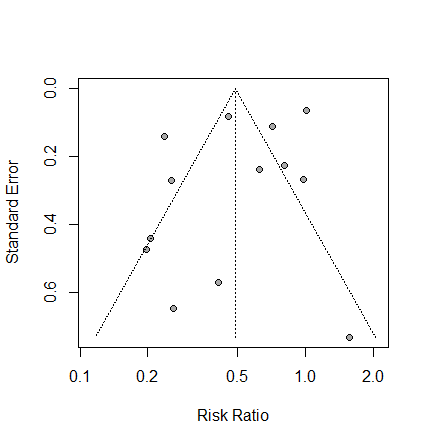
出力されたForest plotは左右が短く、上下の余白が大きめになっているので、R上で大きさを変更してから、右クリックしてコピーしてPPTやWordなどに貼り付けたり、ファイルとして保存します。Forest plotもFunnel plotも、プロットを選択して、ファイルメニューからも別名で保存からPNG、JPEG、PDF、TIFF、Postscript他の形式を選んで、保存することもできます。
なお、Funnel plotの非対称性の統計学的解析法であるEggerの検定、Beggの検定は研究数が10以上無いと実行されません。
metaではRevMan5の形式のForest plotの右側にバイアスリスク評価の結果を含めることもできるそうです。興味のある方はトライしてみて下さい。with-meta-all.Rのスクリプトを見たい場合はこちらです。