Google NotebookLMではテキストファイル、PDFファイル、ウェブページのURL、YouTubeの動画のURLなどをソースとしてアップロードして、それに対してAIで概要を作成したり、音声解説や動画解説を作成したり、レポートを作成したり、インフォグラフィックやスライドを作成したりすることができます。
NotebookLMのソースとしてテキストファイルをアップロードする場合、1ソースあたり単語として最大50万語、200MB、1ノートブックあたり、ソース数50個、ファイルサイズ、単語数は制限なしで、しかもタイプの異なるソースをアップロードすることができます。
今回は、ここに、PubMedから、検索結果をAbstract (text)形式でダウンロードしたテキストファイルをソースとしてアップロードして、SR/MAにおける文献の一次選定をAIでできるか試してみようということです。肝細胞癌患者に対するTamoxifenの生存に対する効果を分析したランダム化比較試験のPICO要約表を作成することをNotebookLMでやってみたところ、あっという間に表が作成できました。
Cochrane systematic reviewのひとつである、Naing C 2024の論文を基準として、用い、CQは「手術不能の肝細胞癌患者にタモキシフェンによる薬物療法はプラセボまたは同等の治療と比べ生存を改善するか?」です。P:肝細胞癌患者、I:タモキシフェン投与、C: プラセボ・無治療・保存的治療のみ、O: 生存
Naing C 2024の論文では文献検索の対象期間は1990年から2024年3月26日になっています。以下の9件の論文が採用されていました。これらと同じ文献をAIが見つけ出して、PICO要約表を作成できるでしょうか?Barbare JC 2005、Chow PK 2002、Liu CL 2000、CLIP Group 2000、Riestra S 1998、Castells A 1995、Martínez Cerezo FJ 1994、Elba S 1994、Farinati F 1990
今回用いた検索式は以下の通りで、CochraneのRCT用の感度最大検索フィルターを用いています。これで、90件の文献が引き出されました。
(carcinoma,hepatocellular[mh] OR hepatocellular carcinoma[tw]) AND (tamoxifen[mh] OR tamoxifen[tw]) AND humans[mh] AND (english[la] OR japanese[la]) AND hasabstract[tw] AND (randomized controlled trial [pt] OR controlled clinical trial [pt] OR randomized [tiab] OR placebo [tiab] OR drug therapy [sh] OR randomly [tiab] OR trial [tiab] OR groups [tiab])検索結果をAbstract (text)形式でダウンロードしたテキストファイルのファイルサイズは、189KBで単語数は約2.6万、文字数は19.4万でした。小さめのファイルで、分割する必要はなく、そのままNotebookLMにソースとしてアップロードできます。(なお、あらかじめこの検索結果に上記の9つの文献が含まれていることは確認しておきました。)
アップロードすると、以下の様な概要がAIによって書き出されますが、これも有用な情報だと思います:
「原典群は、主に進行性肝細胞がん(HCC)に対する治療戦略、特にホルモン療法であるタモキシフェンの役割について広範な臨床的評価とシステマティックレビューの結果を提示しています。かつて、HCC組織におけるホルモン受容体の発現や疾患の男性優位性に基づきタモキシフェンが有望視されましたが、その治療効果については初期の段階から見解が分かれていました。しかし、複数の大規模な無作為化比較試験(RCT)および更新されたコクラントレビューは、切除不能なHCC患者の全生存期間の延長に対するタモキシフェンの効果はほとんど、あるいは全くないと結論づけています。この一貫した否定的証拠に基づき、タモキシフェンや抗アンドロゲン療法はHCCの標準治療とは見なされていません。研究では、タモキシフェンがもし作用するとすれば、エストロゲン受容体とは独立した経路や高用量での使用が関連している可能性が指摘されており、シスプラチンやドキソルビシンなどの他の化学療法も単剤での有効性には限界があることが示されています。さらに、これらの文書は、HCCの予後を評価する際にCLIPスコアなどのステージングシステムが重要な役割を果たすことを強調しています。」
そして、NotebookLMで以下のプロンプトを実行しました。このプロンプトは雛形として、採用基準・除外基準を書き換えて、他の場合でも使えると思います。
ソースのテキストファイルから、以下の採用基準と除外基準に合致する文献を抽出して、Study ID、P、I、C、O、コメント、PMIDの7列からなるテーブルを作成してください。P,I,C,O,コメントは原則日本語で記述してください。研究は年度の新しい順に並べ、Googleスプレッドシートへエクスポートできるテーブルとして提示してください。
Study IDは第一著者の姓のフルスペル+半角スペース+イニシャル+半角スペース+年度を記述してください。
Pの欄は対象者に関する記述(症例数)、Iの欄は介入に関する記述(症例数)、Cの欄は対照の治療に関する記述(症例数)、Oの欄は測定されたアウトカムの内容を記述してください。
コメント欄は介入の効果の概略を記述してください。
PMIDの欄はhttps://pubmed.ncbi.nlm.nih.gov/PMID/のように、クリックしたらPubMedの該当する文献を開けるようなリンクのURLを記述してください。
P, I, C, Oの欄は他の研究との違いが分かる程度の詳細な情報を含めてください。
採用基準:
研究デザインはランダム化比較試験。
対象は肝細胞癌患者。
介入がタモキシフェン。
対照がプラセボあるいは無治療あるいは保存的治療。
生存をアウトカムとして分析。
除外基準:
システマティックレビュー/メタアナリシスの論文は除外する。
対照が肝動脈塞栓療法や化学塞栓療法の研究は除外する。
アウトカムとして生存が分析されていない研究は除外する。結果は、表として提示され、文献のURLも有効で、クリックするとPubMedのその文献のアブストラクトがブラウザで表示されますので、内容を確認するのも簡単にできます。得られた結果はコピーしてGoogleスプレッドシートに貼り付け、最初の2行を削除しタイトルと、文献リストの部分だけにしました。画像として以下に示します。
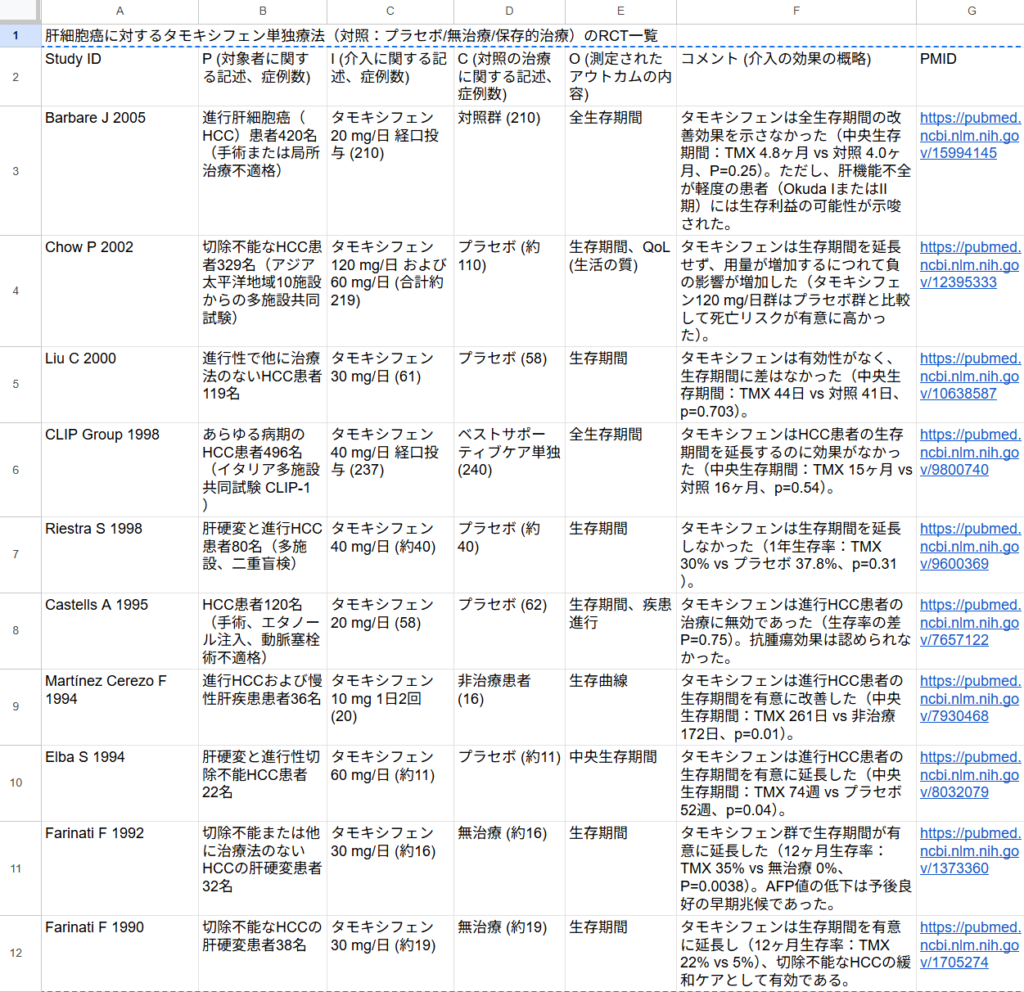
11行目のFarinati F 1992はNaing C 2024の採用論文に含まれていませんでしたが、それ以外はすべて同じ論文が選ばれていました。
この結果からは、SR/MAにおける、文献の一次選定をNotebookLMでやらせれば、簡単に、あっという間にできそうだということになります。
概要を読んで全体を理解し、一次選定した論文の全文を入手して、バイアスリスク評価、非直接性の評価を行い、効果指標の値を抽出し、メタアナリシスを行うわけですが、スピードアップが図れることは間違いないようです。
PubMedのAbstract (text)形式でテキストファイルとしてダウンロードした場合、500件の文献で単語数は15~20万語くらいになり、1000件では約40万語ぐらいになるので、それを超える場合は、ファイルを分割する必要があるでしょう。今回は、90件の文献から10件が選定されましたが、今後、もっと文献数が多い場合も一次選定がうまくできるか試す価値はあると思います。
扱えるコンテキストウインドウの大きさ、トークン(AIの処理する情報の単位、前の投稿を参照)を考えると、GeminiよりもNotebookLMの方がPICO要約表の作成には適していると思います。PubMedでのAbstract (text)形式でテキストファイルとしてダウンロードする方法についても前の投稿を参照してください。

