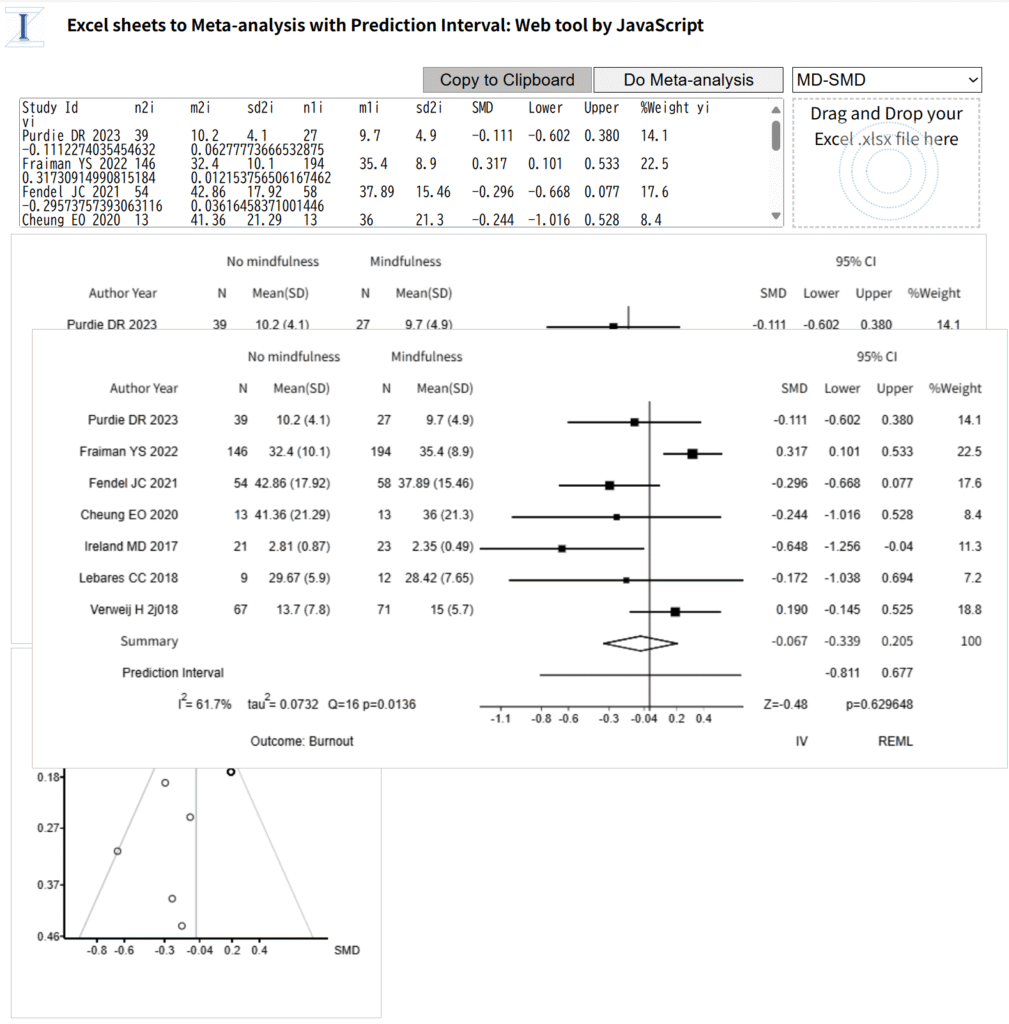
ネットワークメタアナリシスでは、3つ以上の介入あるいは治療の効果を複数の研究からまとめて比較し、直接比較だけでなく間接比較のデータも含めて、各ペアを比較する効果指標の統合値と信頼区間(あるいはは確信区間)および効果の大きさの順位を知ることができます。ペア比較メタアナリシスは二つの介入のうち、どちらを選択すべきかという問題に解答を与えてくれますが、ネットワークメタアナリシスは三つ以上の介入のうちどれを選択すべきか、言い換えると最善の介入はどれかという問題に解答を与えてくれる可能性があります。→参考情報
ネットワークメタアナリシス用のソフトウェアとして、頻度論派メタアナリシスのためにはnetmetaというRのパッケージがありますが、gemtcはベイジアンネットワークメタアナリシスのためのRのパッケージで、rjagsというRのパッケージを介してJAGS (Just Another Gibbs Sampler)でMCMC(Malkov Chain Monte Carlo)シミュレーションを実行させます。JAGSとrjagsはあらかじめインストールしておきます。Rで使用する際には、library(rjags);library(gemtc)を最初に実行する必要があるのは、他のRのパッケージと同じです。
MCMCでは事後分布から、必要に応じて、数万から100万程度のサンプリング値を得るので、実行には時間がかかります。事前分布にはほぼ平坦な分布を用いますが、既知の情報に基づいて設定することもできます。
gemtcでは連続変数アウトカムの場合、平均値差Mean Difference (MD)を効果指標として取り扱えますが、最近、標準化平均値差Standardized Mean Difference (SMD)も取り扱えるようになりました。各研究からアームごとの研究ID、治療、平均値、標準偏差、サンプルサイズのデータを抽出します。列名はstudy, treatment, mean, std.dev, sampleSizeとします。同じ研究内では最初の行が参照アームになります。
このようなデータ形式をArm-level dataと呼んでいます。また、あらかじめ計算した平均値差とその標準誤差、あるいは、標準化平均値差とその標準誤差からもネットワークメタアナリシスを実行することもできます。この場合でも、netmetaなどのデータ形式と異なり、1行1アームの形式になります。列名はstudy, treatment, diff, std.errとなります。このようなデータ形式をRelative effect dataと呼んでいます。
1つの行に2つのアームを比較した相対的効果指標とその標準誤差が含まれているのですが、参照アームの行も必要で、参照アームのdiffの欄はNAとします。2アームだけしかない場合は、std.errの欄もNAで問題ないのですが、3アーム以上ある場合は、参照アームのstd.errには参照アームの標準誤差の値が必要になります。平均値差であれば、対照アームの標準偏差をサンプルサイズの平方根で割り算した値になりますし、標準化平均値差の場合はサンプルサイズの平方根の逆数になります。参照アームの標準誤差の値がNAだとモデル作成時にエラーになります。
通常は、元データとして、各アームの平均値と標準偏差を用意したほうがいいでしょう。一部の研究で例えば、平均値差と標準誤差、あるいは標準化平均値差と標準誤差のデータしか得られないような場合、他の研究について、これらの相対効果指標と標準誤差を計算して解析するような場合は、Relative effect dataを使うことになるでしょう。
gemtcでは最初に、mtc.network()関数でネットワークオブジェクトを作成しますが、Arm-level dataの場合は、netw=mtc.network(data.ab=data)と記述し、Relative effect dataの場合は、netw=mtc.network(data.re=data)と記述します。
mtc.model()関数は変数netwに対して演算処理を行いますが、likelihoodとlinkの引数に効果指標のタイプに応じた値を設定する必要があります。元データが平均値と標準偏差の場合は、likelihood=”normal”, link=”identity”とすると、統合値は平均値差になります。liklihood=”normal”, link=”smd”とすると統合値は標準化平均値差になります。標準化平均値差はHedge’s gが用いられています。Cohen’s dに対し、サンプルサイズが小さい場合のずれを調整する項が追加されています。
元データが、Relative effect dataで、平均値差と標準誤差の場合、likelihood=”normal”, link=”identity”とすると、統合値は平均値差になります。
元データが、Relative effect dataで、標準化平均値差と標準誤差の場合、likelihood=”normal”, link=”identity”とすると、統合値は標準化平均値差になります。ただし、gemtcのforest()関数で、フォレストプロットを作成すると、ラベルがMean Differenceとなりますので、あとで画像として開いて書き換えるか、forestplotパッケージを使うなどして、別の方法でフォレストプロットを作成する必要があります。
例えば、netw = mtc.network(data.ab = data)で作成したネットワークオブジェクトの変数名がnetwだとすると、次のステップではmtc.model()関数で、netwに対してモデル作成を行います。
model <- mtc.model(netw, likelihood = lh, link = lk, linearModel=rndfix, n.chain=chain )
変数lh, lkには上記の文字列を格納します。ランダム効果モデルの場合は、rndfix = “random”とします。chainはMCMCを繰り返す回数です、例えば、chain=4とします。
その後、例えば、以下の様にMCMCを実行させます。
burnin = 10000
iteration = 110000
thin = 1
res <- mtc.run(model, n.adapt = burnin, n.iter = iteration, thin = thin)
以上を解説したPDFのスライドファイルをこちらで閲覧できます(4.3MB)。
gemtcはもちろん、リスク比、リスク差、オッズ比、ハザード比も取り扱えます。元データのフォーマットと、likelihood、linkの設定が分からないと、使えないので、今回特に、連続変数アウトカムの場合について、解説しました。実際のRのスクリプトやMCMCの結果の利用法についてはいつか解説したいと思います。